编者按
目前研究证实,乙型肝炎病毒(HBV)DNA整合是慢性乙肝进展及肝癌发生的关键“隐形推手”之一。需要注意的是,整合DNA作为“基因组印记”稳定存在于宿主细胞中,现有核苷类似物或干扰素治疗虽然能抑制病毒复制,但尚无法彻底清除cccDNA和整合DNA序列,成为实现乙肝临床治愈及完全治愈的核心障碍。近日,在中华医学会第二十二次病毒性肝炎及肝病学术会议暨2025年中华医学会感染病学分会年会、中华医学会肝病学分会年会上,四川大学华西医院感染性疾病中心唐红教授就“乙肝病毒DNA整合的研究进展及临床意义”相关话题进行了精彩的学术报告。本刊特将报告内容整理如下,供读者参阅。

1980年,Nature杂志发表了一项里程碑式研究[1],首次证实HBV DNA可整合到人类肝细胞基因组中,这一发现为揭示HBV的致病机制提供了关键线索。目前,HBV DNA整合已成为全球肝病研究的重要议题之一。最新数据显示,PubMed数据库收录的相关论文已达2602篇,其中中国学者贡献306篇,平均每篇被引用35.33次,展现出我国在该领域的研究深度与国际影响力[2]。
HBV DNA整合的分子机制及特点HBV DNA整合是指HBV的部分或全长基因组片段插入到宿主DNA中的过程,整合的HBV DNA来源于线性双链DNA(dslDNA)。总的来说,乙肝病毒DNA的整合过程遵循“复制错误-修复异常-整合发生”的三步逻辑链(图1)。这一过程始于病毒基因组的复制环节[3]:HBV病毒颗粒进入肝细胞后,松弛环状DNA(rcDNA)被修复形成共价闭合环状DNA(cccDNA),cccDNA转录生成前基因组RNA(pgRNA);以pgRNA作为模板,逆转录合成HBV负链DNA,聚合酶准确转位则进一步合成HBV正链DNA形成松弛环状DNA(rcDNA);如果聚合酶发生错误转位,则会导致线性双链DNA(dslDNA)的产生,而这正是病毒DNA整合入宿主染色体的关键中间体(图2)。

图1. HBV的生活史
(引自讲者幻灯)
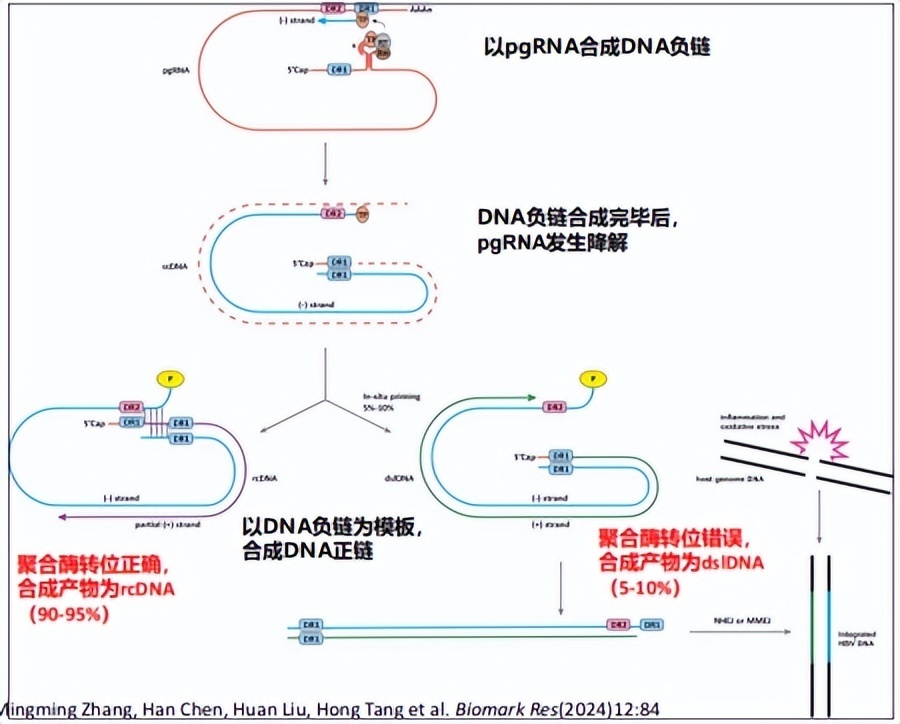
图2. dslDNA的产生
(引自讲者幻灯)
目前较多研究认为,HBV整合的总体分布是随机的。但也有一些研究显示[4-6],基因组上转录活跃的区域由于解旋过程失去了核小体的保护,更容易受到损伤和断裂。病毒整合可能更倾向于发生在基因富集区域,例如CpG岛、基因编码区和启动子区域,而并非完全随机分布(图3)。在HBV基因组中,整合通常发生在DR1或DR2位点附近,特别是在HBx基因的3’端和pre-C/C基因的5’端附近(大约1800处),整合的HBV片段大小范围从28 bp至3215 bp不等,其中长度为1500-2500 bp的片段出现频率明显高于其他长度的片段。
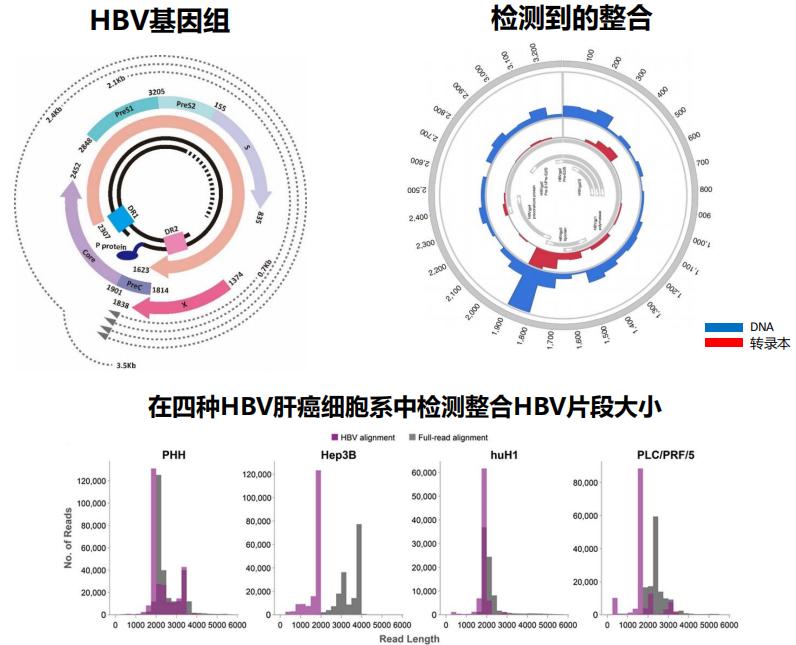
图3. HBV DNA整合在HBV基因组中的特点
(引自讲者幻灯)
当HBV DNA整合侵入宿主基因组时,整合可影响下游宿主基因的表达,当整合不断积累时,细胞会启动一种紧急修复机制。但这种修复如同“盲人拼图”,极易出错——病毒DNA与宿主染色体的断裂末端被错误连接,导致染色体片段断裂、倒位或易位。这些基因组结构的不稳定为后续的细胞病变埋下隐患[4,7]。
此外,病毒DNA整合后,常导致病毒基因的“残缺表达”,生成失去正常功能的截短病毒蛋白,其中截短型HBx过度表达可抑制细胞凋亡[8],不能正确分泌的截短型HBsAg积累在内质网膜,可引起内质网应激和细胞氧化应激,进一步增加DNA损伤和双链断裂,反过来又增加了整合的概率[9]。
总之,HBV DNA整合主通过影响宿主基因的表达、改变染色体的稳定性、产生截短型病毒蛋白等在HCC的发生中产生作用,分析并理解这其中的分子机制具有重要意义。
HBV DNA整合的检测近年来,乙肝病毒DNA整合检测技术经历了从低通量到高通量的跨越式发展(表1)。Southern Blot技术是最早的整合检测方法,但仅能检测高度扩增的克隆肝细胞,灵敏度较低。PCR技术的出现显著提升了检测灵敏度。其中,Alu-PCR利用人类基因组中Alu重复序列(占基因组11%)的分布特征,通过设计病毒特异性引物与Alu序列引物,特异性扩增病毒-宿主连接位点,使检测灵敏度提升至10-4水平。此外,随着全基因组测序(WGS) 技术的应用,改变了整合检测的格局。通过对肿瘤组织进行全基因组测序,研究者可在全基因组范围内无偏倚地识别整合位点,实现“一网打尽”。
表1. HBV DNA整合的检测手段
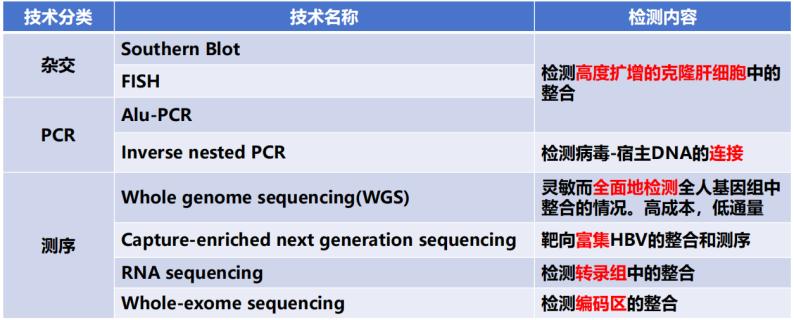
(引自讲者幻灯)
注:高度扩增的克隆肝细胞,是指某些肝细胞经历了异常的增殖过程,形成了数量异常增多的细胞群体(克隆群),常与病毒整合及癌变相关。
近年来,在探索HBV DNA整合的无创生物标志物过程中发现,肝癌和肝硬化患者血浆中的循环cfDNA中可检测到HBV整合,这种携带整合的cfDNA称为vh-DNA[10]。由于cfDNA主要来源于死亡的肿瘤细胞,非肝癌肝组织释放的cfDNA显著低于肝癌组织。因此,vh-DNA可用于监测肝癌进展过程中的HBV整合,也可以作为监测残余肿瘤细胞和预测复发的生物标志物(图4)。
研究显示,在肝癌患者中,血浆vh-DNA的检出率高达83%,而在肝硬化患者中仅为21%。这一显著差异提示,vh-DNA有望成为区分肝癌与良性肝病的精准生物标志物,帮助更早识别高风险人群[11-13]。
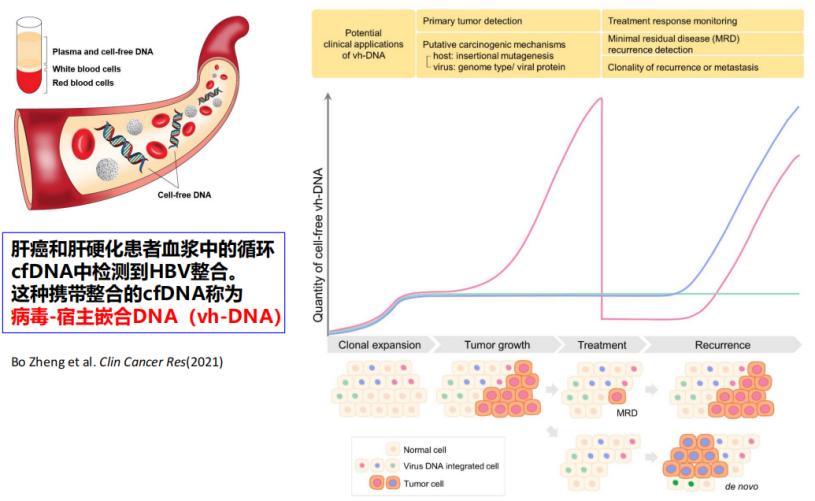
图4. vh-DNA监测残余肿瘤细胞和预测复发的效果评估
(引自讲者幻灯)
HBV DNA整合的临床意义HBV感染过程中,病毒DNA整合并非静态事件,而是随着病毒复制活性与宿主免疫压力的变化呈现动态演变,不同感染阶段呈现出不同的整合特征(图5)。整合在HBV感染早期即可发生,HBV整合数量与肝脏炎症呈正相关,在HBsAg阴转的情况下,整合仍然可以存在。在感染早期,病毒基因组即可突破免疫防线,通过非同源末端连接等机制插入人体染色体,这种"早潜伏"特性使得慢性乙肝患者在疾病初期就埋下癌变隐患。整合后的病毒DNA片段常发生截短或重排,表达如HBx截短蛋白等异常产物,通过激活癌基因、抑制抑癌通路等方式持续驱动细胞恶性转化,这正是部分患者在病毒载量极低情况下仍发生肝癌的重要原因之一(图6)。
整合的S序列是HBsAg的来源之一,表达整合来源HBsAg的克隆性扩增肝细胞的数量随年龄增长而增加,但机体内HBsAb特异性T淋巴细胞的数量则随年龄增长而减少[14-16]。
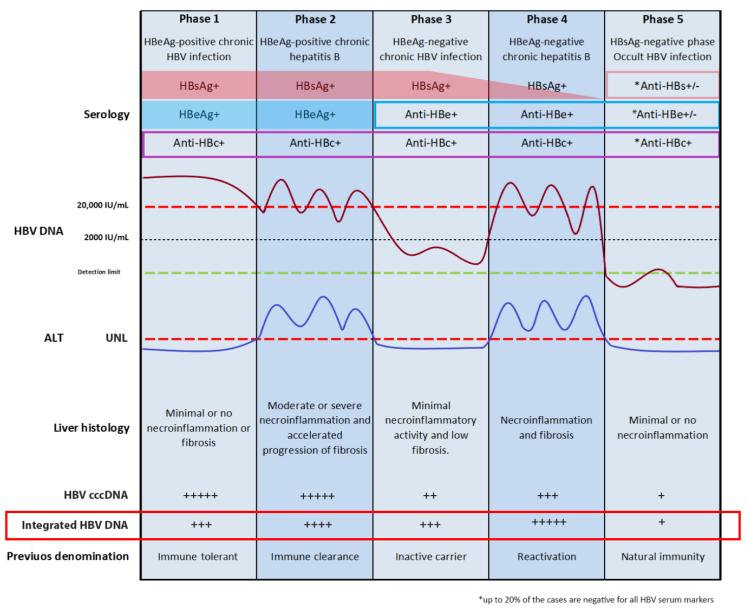
图5. 不同HBV感染阶段中整合的变化
(引自讲者幻灯)

图6. HBV DNA整合是HBV-HCC发生的重要原因之一
(引自讲者幻灯)
多项研究表明[17-19],抗病毒治疗可通过抑制病毒复制,减少HBV整合事件的数量。特别是长期抗病毒治疗,整合事件的数量显著下降。整体看抗病毒治疗使患者获益的分子机制主要包括:减少dslDNA的产生;缓解肝组织局部炎症反应,减轻氧化损伤,减少宿主双链DNA断裂,降低整合发生的概率;减少含有HBV整合的肝细胞的选择性扩增(图7)。
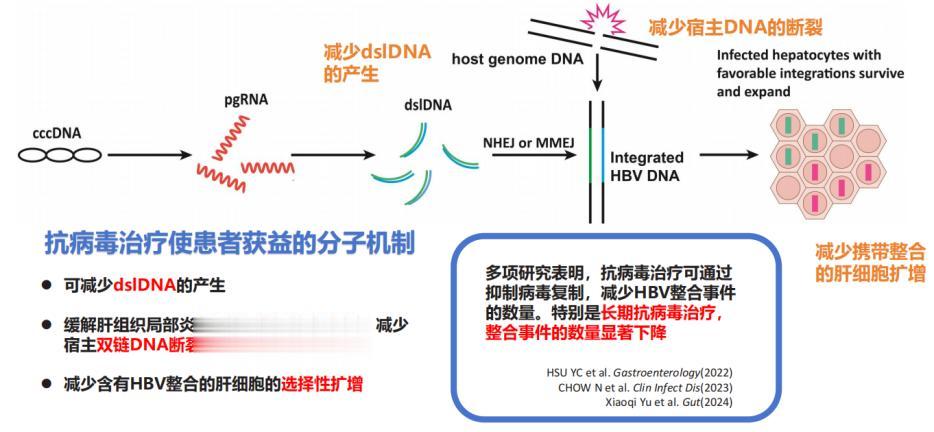
图7. 抗病毒治疗能抑制整合
(引自讲者幻灯)
当前针对HBV DNA整合的治疗策略各有侧重,既有临床验证的成熟方案(如抗病毒药物治疗),也有充满潜力的前沿技术,共同勾勒出乙肝治愈的可能路径。
核苷类药物(NAs)NAs的作用主要为抑制HBV DNA聚合酶功能,减少病毒复制,进而减少新的HBV DNA整合。中国台湾学者的一项研究显示[20],接受NAs治疗3年的患者,肝细胞内HBV整合序列数量平均下降67%,特别是转录活跃的HBV DNA数量显著下降,有效延缓了整合相关肝损伤的进展(图8)。不过,这类药物只能减少新整合,无法清除已经整合到宿主基因组中的病毒序列。
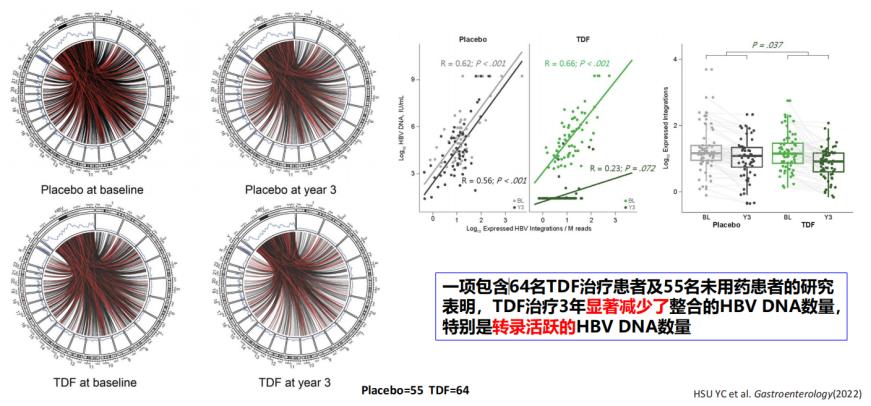
图8. 核苷类药物的治疗能抑制HBV DNA整合
(引自讲者幻灯)
干扰素-α(IFN-α)IFN-α可通过减少pgRNA的合成减少新的整合,同时可增强宿主免疫反应,清除携带整合的克隆肝细胞。PEG IFNα不仅可以抑制病毒复制,其对患者还有免疫调节作用,能够通过激活HBV特异性CD8+ T细胞,甚至清除既存的携带整合DNA片段的克隆性扩增肝细胞[21]。2023年发表的一项研究证实[22],在实现临床治愈的乙肝患者中,肝细胞内整合的HBV DNA数量显著低于未治愈患者,提示IFN-α可能通过增强免疫系统的识别-清除能力,降低整合克隆的存活概率(图9)。

图9. 功能性治愈对HBV DNA整合的影响
(引自讲者幻灯)
此外,还有一些正在研究中的能消除HBV DNA整合的消除策略[23-29](表2)。比如,近年来备受关注的基因编辑技术(如CRISPR/Cas9),通过设计特异性sgRNA,直接靶向切割已整合的HBV DNA序列。
表2. 研究中的能消除整合的治疗策略

(引自讲者幻灯)
总结与展望HBV DNA整合在感染早期即可出现,并贯穿感染全程,是肝癌发生的重要因素之一。通过促进癌症相关基因表达、增加染色体不稳定性及表达截短型HBV蛋白等机制,促进肝癌发生发展。近年来,测序相关的先进的检测技术提升了检测HBV整合的能力,为临床研究提供了重要支持。
抗病毒治疗能够减少整合事件,早期抗病毒治疗具有重要意义。但目前的抗病毒药物尚难以完全清除整合DNA,cccDNA和HBV DNA整合在“临床治愈”后仍可存在,成为HBV再激活及HCC发生的重要风险因素。针对HBV整合的治疗策略正在不断探索,正在成为乙肝治愈和肝癌防治新的研究方向。
参考文献:
1.Brechot C et al.Nature(1980).
2.Jia Liu et al. Hepatic Medicine: Evidence and Research(2024).
3.Mingming Zhang, Han Chen, Huan Liu, Hong Tang et al. Biomark Res(2024)12:84.
4.Ling-Hao Zhao et al,Nature Communications(2016).
5.Wing-kin Sung et al,Nature Genetics(2012).
6.Pierrick Moreau et al,Nature Communications(2018).
7.Zhaoshi Jiang et al,Genome Res(2012).
8.Ning-Fang Ma et al.Clin Cancer Res(2008).
9.Romina Salpini et al.Frontiers in Microbiology(2008).
10.Bo Zheng et al. Clin Cancer Res(2021).
11.Wei Chen et al.Hepatol int(2020).
12.Li CL et al.Hepatol Dec(2020).
13.Li CL et al.Cancers(2022).
14.Teresa Pollicino et al, Viruses(2021).
15.Wooddell Cl et al, Sci Transl Med(2017).
16.William S.Mason et al, Gastroenterology(2016).
17.HSU YC et al. Gastroenterology(2022).
18.CHOW N et al. Clin Infect Dis(2023).
19.Xiaoqi Yu et al. Gut(2024).
20.HSU YC et al. Gastroenterology(2022).
21.周召, 等. 临床肝胆病杂志, 2023, 39(01): 31-36.
22.Na Gao et al, Aliment Pharmacol Ther(2023).
23.Cradick TJ et al, Mol Ther May(2010)
24.Weber ND et al, Mol Ther(2013)
25.Seeger C et al, Mol Ther Nucleic Acids Dec(2014)
26.Wooddell CI et al, Viruses(2021)
27.Smith T et al, Viruses(2021)
28.Gao L et al, Front Immunol(2022)
29.Zhang J et al, Journal of Medical Virology(2023)